De Novo輔助的DIA同源序列檢索提升低豐度突變肽的鑒定
數(shù)據(jù)非依賴采集(DIA)可通過劃分采集窗口,將所有檢測范圍內(nèi)的離子進(jìn)行碎裂和掃描,定量準(zhǔn)確性高、重現(xiàn)性好、蛋白質(zhì)組覆蓋深度廣,尤其適合低豐度肽段的檢測。但現(xiàn)有DIA數(shù)據(jù)分析方法主要依賴譜圖庫或直接數(shù)據(jù)庫搜索,但兩者均受限于數(shù)據(jù)庫中的已知肽段列表,難以發(fā)現(xiàn)由遺傳變異或突變產(chǎn)生的新生肽段。2025年5月30日,Bioinformatics Solutions Inc.發(fā)表了最新預(yù)印本文章 [1],介紹了一種直接從復(fù)雜的DIA譜圖中發(fā)現(xiàn)低豐度突變肽段的算法,同時嚴(yán)格控制FDR。
DIAVariants工作流程
如圖1所示,讀取數(shù)據(jù)后,首先進(jìn)行MS1特征峰提取,以識別電離肽段產(chǎn)生的具有明確同位素的可靠MS1信號響應(yīng)。接下來進(jìn)行PEAKS DIA數(shù)據(jù)庫搜索,將譜圖與參考蛋白質(zhì)序列庫比對,篩選FDR小于1%的可信肽段,并基于保留時間和母離子 m/z與MS1特征峰關(guān)聯(lián)。
對數(shù)據(jù)庫匹配不可信的MS1特征峰,利用DIA de novo預(yù)測與關(guān)聯(lián)MS2最佳匹配的肽段序列。然后通過SPIDER算法,基于de novo預(yù)測的序列、MS1特征峰信息和參考序列校正測序錯誤。SPIDER候選肽段中,僅保留包含1-2個突變位點(diǎn)的序列,下一步預(yù)測這些候選突變肽的索引保留時間(iRT),通過RT回歸模型轉(zhuǎn)換為實(shí)驗(yàn)RT,過濾掉預(yù)測iRT與實(shí)測RT差異顯著的突變肽段。最后,將候選突變肽與數(shù)據(jù)庫搜索得到的肽段合并構(gòu)建臨時譜圖庫,基于實(shí)測RT進(jìn)行PEAKS DIA譜圖庫搜索,通過Q值控制FDR。
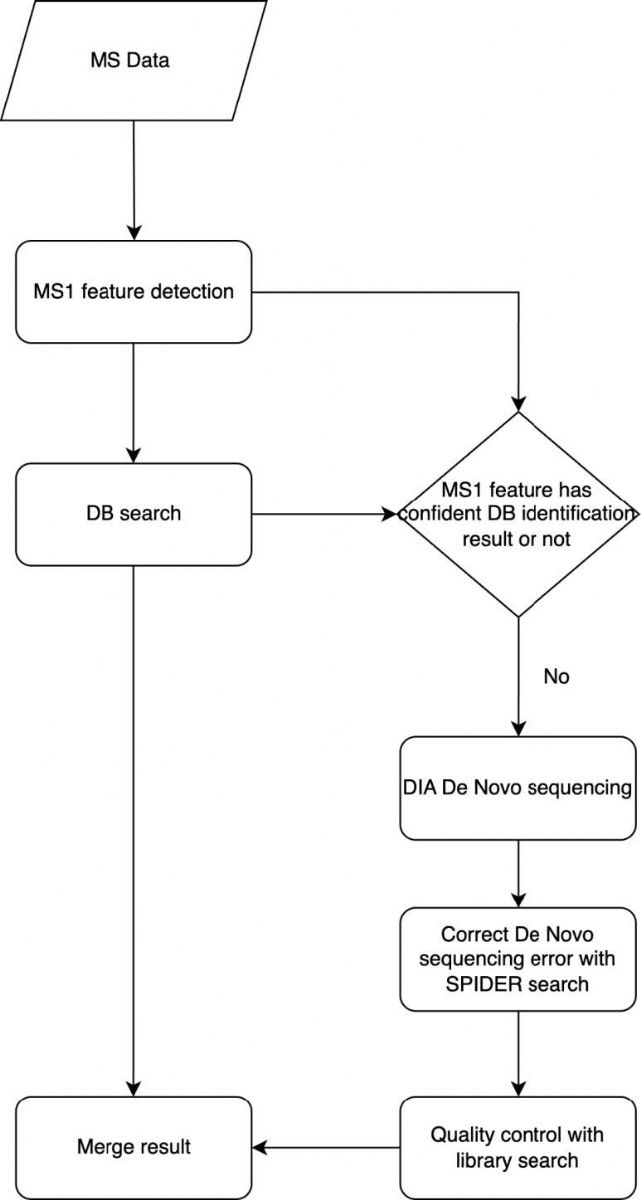
圖1 DIAVariant方法流程
結(jié)果展示PEAKS DIA直接數(shù)據(jù)庫搜索性能
下載兩個公開質(zhì)譜數(shù)據(jù)集(PXD046453、PXD050030),采用相同的database和參數(shù),分別使用PEAKS DIA DB search和DIANN2.1進(jìn)行分析,結(jié)果如表1所示,從定量蛋白數(shù)量、CV指標(biāo)來看,兩者在DIA蛋白質(zhì)組數(shù)據(jù)的定量性能上,穩(wěn)定性和靈敏度均較高。
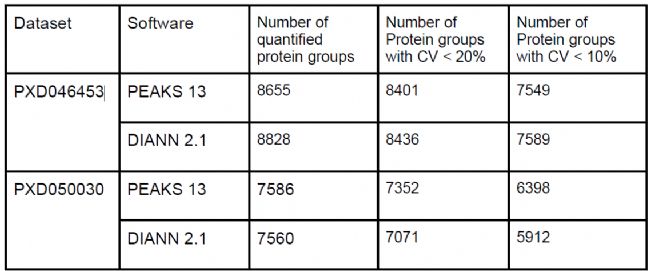
表1 文獻(xiàn)數(shù)據(jù)分析結(jié)果對比
跨物種搜索驗(yàn)證DIAVariants特異性
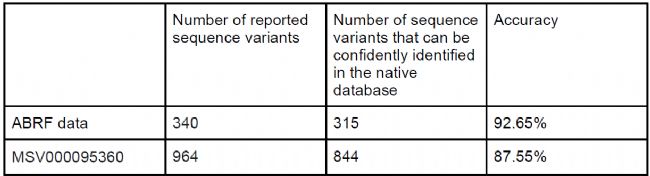
在ABRF人類樣本(搜索小鼠數(shù)據(jù)庫)和MSV000095360小鼠樣本(搜索人類數(shù)據(jù)庫)的結(jié)果中,DIAVariant報告的肽段變體中85%以上可在本物種數(shù)據(jù)庫中被可信鑒定(q-value <0.01),驗(yàn)證了算法的高特異性(表2)。
與蛋白基因組方法對比
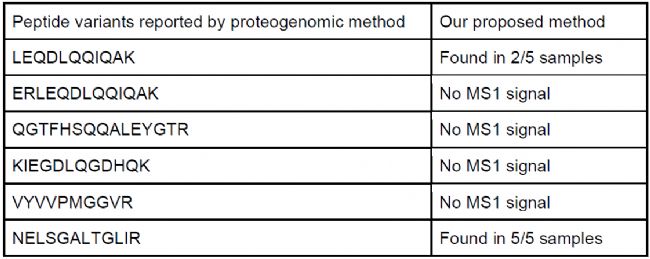
Fierro-Monti等[2]曾對Hela細(xì)胞樣本進(jìn)行外顯子組測序,通過蛋白基因組的方法構(gòu)建了233個經(jīng)典蛋白變體,將其加入人類參考序列數(shù)據(jù)庫后,使用DIANN 1.8.1檢索到了相應(yīng)的DIA數(shù)據(jù),并通過同位素合成肽段和靶向驗(yàn)證,最終報告了6個肽段變體。我們下載了文獻(xiàn)中的原始數(shù)據(jù),用DIAVariants直接分析,結(jié)果成功鑒定到了上述6條肽段變體的其中2條(LEQDLQQIQAK 和 NELSGALTGLIR)(表 3),并且額外發(fā)現(xiàn)426個低豐度潛在多肽變體。這些低豐度肽段因MS1信號弱,難以通過DDA數(shù)據(jù)檢測,更加顯示了DIA-MS在低豐度肽段檢測中的優(yōu)勢。
DIAVariant通過整合DIA數(shù)據(jù)庫搜索、從頭測序和同源校正,提供了一種高效、無偏的DIA數(shù)據(jù)突變肽段識別方法,可同時檢測參考數(shù)據(jù)庫內(nèi)的已知肽段和數(shù)據(jù)庫外的肽段變體,尤其適用于低豐度肽段和復(fù)雜遺傳變異的分析。該方法為蛋白質(zhì)組學(xué)中序列變體的發(fā)現(xiàn)提供了新工具,有望推動精準(zhǔn)醫(yī)學(xué)和癌癥新抗原研究的發(fā)展。但目前僅驗(yàn)證了1-2個氨基酸突變位點(diǎn),未涉及插入和缺失的情況,未來算法仍會繼續(xù)擴(kuò)展。
參考文獻(xiàn)
[1] Qiao, R., et al. (2025). "De Novo sequencing-assisted homology search for DIA data analysis enables low abundance peptide variants discovery." doi: https://doi.org/10.1101/2025.05.30.657054.
[2] Fierro-Monti, Ivo, et al. "Assessment of Data-Independent Acquisition Mass Spectrometry (DIA-MS) for the Identification of Single Amino Acid Variants." Proteomes 12.4 (2024): 33.
原文鏈接:https://www.biorxiv.org/content/10.1101/2025.05.30.657054v1
作為生物信息學(xué)的領(lǐng)軍企業(yè),BSI專注于蛋白質(zhì)組學(xué)和生物藥領(lǐng)域,通過機(jī)器學(xué)習(xí)和先進(jìn)算法提供世界領(lǐng)先的質(zhì)譜數(shù)據(jù)分析軟件和蛋白質(zhì)組學(xué)服務(wù)解決方案,以推進(jìn)生物學(xué)研究和藥物發(fā)現(xiàn)。我們通過基于AI的計算方案,為您提供對蛋白質(zhì)組學(xué)、基因組學(xué)和醫(yī)學(xué)的卓越洞見。旗下著名的PEAKS®️系列軟件在全世界擁有數(shù)千家學(xué)術(shù)和工業(yè)用戶,包括:PEAKS®️ Studio,PEAKS®️ Online,PEAKS®️ GlycanFinder, PEAKS®️ AB及抗體綜合表征服務(wù)等。
聯(lián)系方式:021-60919891;sales-china@bioinfor.com
- De Novo輔助的DIA同源序列檢索提升低豐度突變肽的鑒定
- 用戶文章:結(jié)合質(zhì)譜蛋白質(zhì)測序解碼蛋白質(zhì)糖基化
- 蛋白質(zhì)組學(xué)基礎(chǔ): 質(zhì)譜中肽段主要碎裂方式
- 蛋白質(zhì)組學(xué)基礎(chǔ) :常見蛋白酶的結(jié)構(gòu)、來源及性質(zhì)
- 用戶文章:食品加工使半夏中毒性凝集素減少和生物活性肽增加
- 針對SARS-CoV-2變異株核衣殼蛋白的單克隆抗體的開發(fā)與表征
- DeepSearch——基于深度學(xué)習(xí)的高靈敏串聯(lián)質(zhì)譜數(shù)據(jù)搜庫分析策略
- 國內(nèi)常見的16款電子實(shí)驗(yàn)記錄本ELN簡介