三代全長16S/18S/ITS擴(kuò)增子測序的介紹
三代微生物多樣性是基于PacBio SequelⅡ測序平臺,利用單分子實(shí)時測序的方法,對原核生物16S的全部V1-V9可變區(qū)域或真核生物的18S高變區(qū)域或ITS區(qū)域進(jìn)行全長擴(kuò)增,不僅能提高物種鑒定的分辨率,還能提高樣本中微生物組成鑒定的精確度,從而更全面的反應(yīng)微生物的群落結(jié)構(gòu)。
一、PacBio測序原理
利用與二代測序相同的邊合成邊測序的原理,以SMRT芯片為載體;模板與特殊聚合酶結(jié)合后,4種不同熒光標(biāo)記的脫氧核糖核苷三磷酸(dNTP)通過布朗運(yùn)動隨機(jī)進(jìn)入檢測區(qū)與聚合酶結(jié)合,按DNA模板序列延伸;通過檢測單個DNA分子的合成所產(chǎn)生的信號來進(jìn)行測序,也可通過檢測相鄰兩個堿基的測序時間,來檢測一些堿基修飾情況。
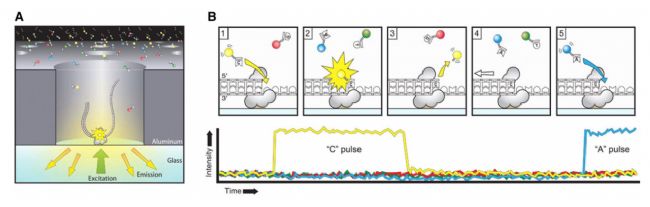
圖1. PacBio測序原理[1]
二、產(chǎn)品優(yōu)勢于二代測序只能獲取1~2個高變區(qū)短讀長序列而言,三代測序能一次性測到所有高變區(qū),同時也可以避免不同引物帶來的偏好性,從而提高微生物群落的分類學(xué)分辨率。
1.更長讀長,更高準(zhǔn)確率[2]

圖2. 測序長度和準(zhǔn)確度
2.更高的物種鑒定分辨率和精確度[3]
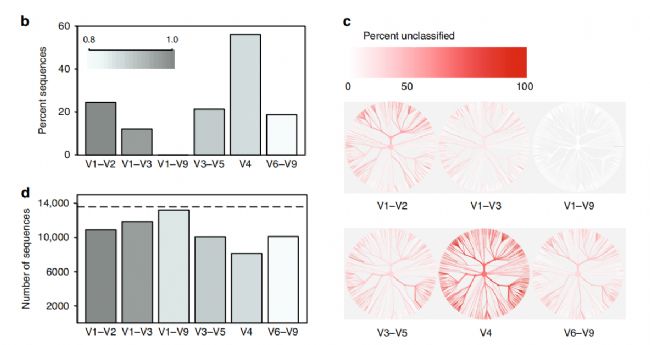
圖3. 物種鑒定結(jié)果
3.更準(zhǔn)確的還原微生物群落[4]
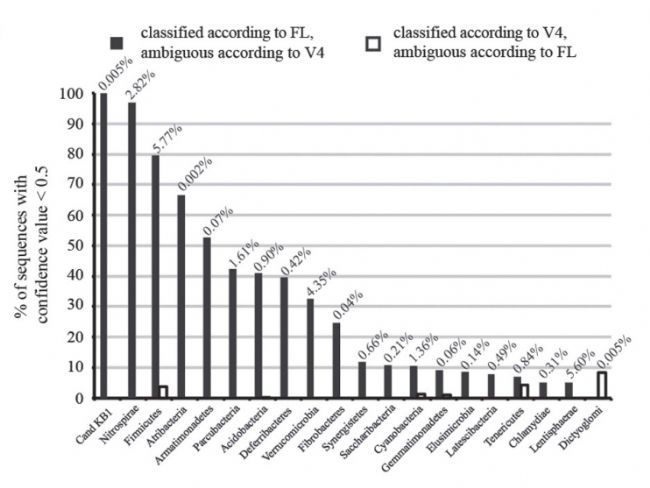
圖4. 微生物群落分布
三、技術(shù)路線
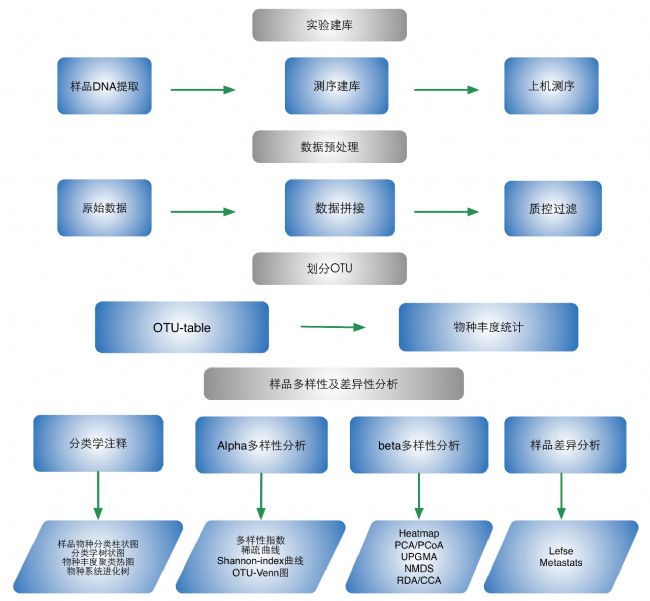
圖5. 技術(shù)流程圖
四、案例分享文章標(biāo)題:三代全長微生物多樣性研究母嬰微生物群[5]
研究小結(jié):早期微生物與成年后疾病的發(fā)生發(fā)展有一定的關(guān)系,研究發(fā)現(xiàn)子宮、胎便、羊水和胎盤都有微生物存在,那么嬰兒胎便微生物來源與母體不同來源的樣本的關(guān)系如何呢?本文以39對母嬰的產(chǎn)婦樣本(羊水、糞便、陰道分泌物和唾液)和嬰兒胎便樣品為研究對象,基于PacBio三代全長測序技術(shù)進(jìn)行16S rRNA檢測。檢測發(fā)現(xiàn)不同類型樣本間α多樣性和β多樣性存在特異性;同時除發(fā)現(xiàn)不同類型樣本各自的一些優(yōu)勢菌(其中羊水和陰道分泌物的優(yōu)勢菌為Lactobacillus和Curvibacter,胎便的優(yōu)勢菌為Bacillus 和Escherichia/Shigella,產(chǎn)婦糞便的優(yōu)勢菌為Bacteroides和Faecalibacterium,產(chǎn)婦唾液的優(yōu)勢菌為Streptococcus和Prevotella)外,還發(fā)現(xiàn)了他們間共有OTUs,其中胎便菌群與羊水菌群的共有OTUs多于產(chǎn)婦糞便菌群和陰道菌群,由此推斷羊水菌群相比其他位點(diǎn)貢獻(xiàn)更大。此外,發(fā)現(xiàn)剖腹產(chǎn)和順產(chǎn)胎兒的胎便和羊水微生物群落結(jié)構(gòu)無顯著差異。
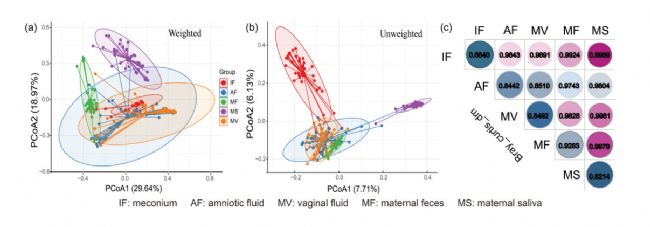
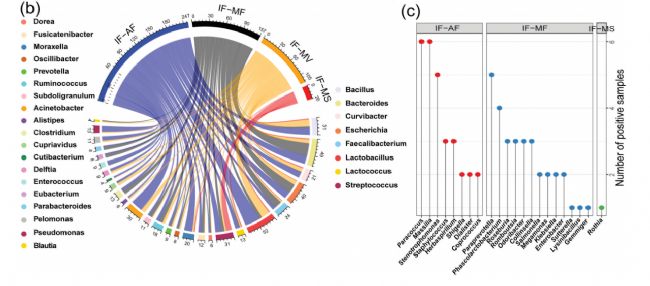
圖6. 文章部分結(jié)果
參考文獻(xiàn):
[1] Mohit K Midha, Mengchu Wu , Kuo-Ping Chiu. Long-read sequencing in deciphering human genetics to a greater depth. Hum Genet . 2019 Dec;138(11-12):1201-1215.[2]PacBio. 2022. PacBio Sequel Systems-PacBio.[online]Availableat: [Accessed 14 February 2022].
[3] Jethro S Johnson, et al. Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis. Nat Commun. 2019 Nov 6;10(1):5029.
[4] Esther Singer, et al. High-resolution phylogenetic microbial community profiling. ISME J.2016 Aug;10(8):2020-32.
[5] Qiuwen He, et al. The meconium microbiota shares more features with the amniotic fluid microbiota than the maternal fecal and vaginal microbiota. Gut Microbes. 2020 Nov 9;12(1):1794266.
文獻(xiàn)下載鏈接:
https://pan.baidu.com/s/1eNadtYArpOiFy3SUPpPrjQ
提取碼:YG45
標(biāo)簽:
代謝組學(xué)
- DNA/RNA聯(lián)檢在生物醫(yī)藥領(lǐng)域生物制品中支原體檢測方向的分析
- 高分辨率空間轉(zhuǎn)錄組技術(shù)揭示胰腺癌轉(zhuǎn)移過程中的譜系塑性
- 免疫與癌癥治療的關(guān)鍵靶點(diǎn)CXCR4的結(jié)構(gòu)功能及相關(guān)實(shí)驗(yàn)介紹
- 疾病治療的新興靶點(diǎn)EP4受體結(jié)構(gòu)與功能介紹及相關(guān)藥物研發(fā)的應(yīng)用
- 支原體PCR檢測技術(shù):高效精準(zhǔn)的污染防控解決方案
- 細(xì)胞STR鑒定[Frequently Asked Questions(常見問題解答)]
- 單細(xì)胞轉(zhuǎn)錄組+PDX模型揭示膠質(zhì)母細(xì)胞瘤復(fù)發(fā)機(jī)制
- 芯空一號實(shí)現(xiàn)的單細(xì)胞空間轉(zhuǎn)錄組檢測數(shù)據(jù)
Copyright(C) 1998-2025 生物器材網(wǎng) 電話:021-64166852;13621656896 E-mail:info@bio-equip.com